doi: 10.56294/saludcyt20241064
REVISIÓN BIBLIOGRÁFICA
Current Perspectives on Antifungal Resistance: Focus on Opportunistic Mycoses
Perspectivas Actuales Sobre la Resistencia Antifúngica: Enfoque en Micosis Oportunistas
Natalia Raquel Aguiar Muñoz1 ![]() *, Carlos Fernando Yauli Flores1,2
*, Carlos Fernando Yauli Flores1,2 ![]() *
*
1Universidad Técnica de Ambato. Facultad de Ciencias de la Salud, Carrera de Laboratorio Clínico. Ambato, Ecuador.
Citar como: Aguiar Muñoz NR, Yauli Flores CF. Perspectivas Actuales Sobre la Resistencia Antifúngica: Enfoque en Micosis Oportunistas. Salud, Ciencia y Tecnología. 2024; 4:1064. https://doi.org/10.56294/saludcyt20241064
Recibido: 06-02-2024 Revisado: 09-04-2024 Aceptado: 13-06-2024 Publicado: 14-06-2024
Editor: Dr.
William Castillo-González ![]()
ABSTRACT
Introduction: opportunistic mycoses, caused by fungi in individuals with compromised immune systems, are on the rise, posing significant challenges in healthcare. This article reviews antifungal resistance with the aim of highlighting its increasing incidence and the need for new therapeutic strategies and precise diagnostic methods. The necessity for standardized laboratory protocols and the implementation of sensitive techniques for early detection and optimal treatment is emphasized.
Method: a systematic review was conducted using PubMed, Scopus, and Google Scholar; search terms and Boolean operators such as “(Opportunistic Mycoses) AND (Antifungal Resistance)” and “(Antifungals) AND (Resistance Genes)” were used. Additionally, the guidelines established by the PRISMA statement were followed, and the PICO approach was employed.
Results: twelve studies were evaluated; 58,33 % (n=7) assessed antifungal resistance, while 41,67 % (n=5) addressed aspects such as mechanisms of action and genetics. 66 % of the studies evaluated resistance in Candida spp., while 16,7 % analyzed resistance in Aspergillus spp., making them the most studied pathogens. Various resistance genes were evaluated in all studies; moreover, fluconazole was found to be the most studied antifungal, followed by amphotericin B and voriconazole.
Conclusion: early identification of antifungal resistance, the development of new therapies, and the standardization of diagnostic methods are essential to mitigate its impact on public health.
Keywords: Opportunistic Mycosis; Antifungal Resistance; Antifungals; Resistance Genes.
RESUMEN
Introducción: las micosis oportunistas, causadas por hongos en individuos con sistemas inmunológicos comprometidos, están en aumento, lo que plantea desafíos significativos en la atención médica. Este artículo revisa la resistencia antifúngica con el objetivo de destacar su creciente incidencia y la necesidad de nuevas estrategias terapéuticas y métodos de diagnóstico precisos. Se subraya la necesidad de protocolos de laboratorio estandarizados y la implementación de técnicas sensibles para una detección temprana y un tratamiento óptimo.
Método: se realizó una revisión sistemática en PubMed, Scopus y Google Scholar; se utilizaron términos de búsqueda y operadores booleanos tales como “(Opportunistic Mycoses) AND (Antifungal Resistance)” y “(Antifungals) AND (Resistance Genes)”. A su vez, se siguieron las directrices establecidas por la declaración PRISMA y se empleó el enfoque PICO.
Resultados: se evaluaron 12 estudios, 58,33 % (n=7) evaluaron la resistencia antifúngica, mientras que el 41,67 % (n=5) abordaron aspectos como mecanismos de acción y genética. El 66 % de los estudios evaluaron la resistencia de especies de Candida spp., mientras que el 16,7 % analizaron resistencias de Aspergillus spp, resultando en los patógenos más estudiados. Fueron evaluados diversos genes de resistencia en todos los estudios; además se encontró que el fluconazol es el antifúngico más estudiado, seguido de la anfotericina B y el voriconazol.
Conclusión: la identificación temprana de la resistencia antifúngica, el desarrollo de nuevas terapias y la estandarización de métodos diagnósticos son esenciales para mitigar su impacto en la salud pública.
Palabras clave: Micosis Oportunistas; Resistencia Antifúngica; Antifúngicos; Genes de Resistencia.
INTRODUCCIÓN
Las micosis oportunistas son infecciones desencadenadas por hongos, ya sean levaduriformes o filamentosos. En condiciones normales estos hongos no representan una amenaza, en pacientes inmunocomprometidos pueden desencadenar infecciones.
Entre las micosis oportunistas más reconocidas se encuentra la aspergilosis, causada por hongos del género Aspergillus spp., la mucormicosis, abarcando especies de los géneros Mucor spp. y Rhizopus spp., y la candidiasis, provocada por hongos del género Candida spp; a su vez, en los últimos años han surgido micosis oportunistas provocadas por hongos poco comunes, entre estas se destacan la faeohifomicosis y la hialohifomicosis.(1)
Las infecciones fúngicas, aunque a menudo se consideran problemas menores, afectan aproximadamente al 15 % de la población mundial de manera superficial. Recientes evidencias muestran un evidente aumento en micosis oportunistas, desencadenando un incremento relevante de patologías graves y riesgo de mortalidad, principalmente entre individuos inmunocomprometidos o con padecimientos subyacentes. Esta proliferación de hongos oportunistas, plantea la importancia de una vigilancia activa y una gestión eficaz de estas infecciones en entornos clínicos.(2)
La resistencia a los antifúngicos es el resultado de una serie de factores, como cambios genéticos específicos en el objetivo celular, el aumento en la producción de las moléculas diana y la expulsión activa de antifúngicos a través de sistemas de bombeo de salida.(3)
La anfotericina B es el principal fármaco antifúngico para tratar infecciones fúngicas invasivas. Este medicamento ha demostrado su eficacia contra una amplia variedad de hongos patógenos, y son pocos los que desarrollan resistencia tras su uso. La introducción de los antifúngicos triazoles, como el itraconazol y el fluconazol, ha ampliado opciones terapéuticas al proporcionar alternativas con menos efectos secundarios.(4)
Los hongos emplean diversas estrategias para fortalecer la resistencia frente a los antifúngicos. Estas estructuras multicelulares generan mecanismos que incluyen el aumento de la densidad celular, la restricción del suministro de nutrientes, el mantenimiento de células persistentes, la activación de genes de resistencia y el incremento del contenido de esteroles en las membranas celulares. Además, la matriz extracelular, compuesta por proteínas, carbohidratos, lípidos y ADN, contribuye a esta resistencia.(5)
Investigaciones recientes han destacado los componentes mitocondriales como posibles objetivos terapéuticos para nuevos antifúngicos, dado su papel crucial en la viabilidad y patogenicidad de los hongos. Se ha observado que albergan más de 1 000 proteínas, la mayoría codificadas por el núcleo celular y altamente conservadas entre distintas especies. Este descubrimiento plantea un desafío en la búsqueda de nuevos agentes antifúngicos dirigidos a las mitocondrias, considerando la estrecha relación evolutiva entre hongos y humanos.(6)
En la práctica clínica contemporánea, el diagnóstico exacto de las resistencias antifúngicas es crucial debido al creciente incremento de infecciones que muestran resistencia a los tratamientos habituales. La detección temprana de la resistencia antifúngica posibilita una intervención oportuna, previniendo complicaciones y mejorando los desenlaces para el paciente.
Además, una evaluación precisa ayuda a prevenir la utilización innecesaria o inefectiva de antifúngicos, disminuyendo el riesgo de efectos secundarios y la presión selectiva que promueve el surgimiento de resistencias. Por ende, el objetivo de este artículo radica en identificar los progresos en la comprensión de la resistencia antifúngica en infecciones causadas por micosis oportunistas y su diagnóstico mediante pruebas de laboratorio específicas.
MÉTODO
Se realizó una revisión sistemática de la literatura, siguiendo un enfoque descriptivo y no participativo, en el periodo de febrero a mayo de 2024 siguiendo la metodología PRISMA y empleando el enfoque PICO.
La población de interés comprendió hongos patógenos que muestran evidencia de resistencia. La intervención se centró en la determinación de la resistencia antifúngica. La comparación se orientó hacia la prevalencia de micosis superficiales versus profundas, y los resultados se enfocaron en la prevalencia de la resistencia antifúngica y los genes asociados.
En base al proceso, de planteó la pregunta de investigación: ¿Cuáles son los avances en la comprensión de la resistencia antifúngica en infecciones por micosis oportunistas y cuáles son las pruebas de laboratorio específicas utilizadas para su diagnóstico?
Se consultaron las bases de datos PubMed, Scopus y Google Scholar, al mismo tiempo, publicaciones especializadas relacionadas con la resistencia antifúngica y las micosis oportunistas, como el Journal of Antimicrobial Chemotherapy, Medical Mycology, Clinical Infectious Diseases, entre otras.
Además, se recurrió a información proporcionada por organismos internacionales como la Organización Mundial de la Salud (OMS), así como a documentos y directrices emitidas por sociedades científicas relevantes en el ámbito de la micología médica.
Se buscó documentos publicado en los últimos 5 años en idioma ingles y de acceso libre, que tratan sobre las micosis que afectan a los seres humanos y que expliquen los mecanismos relacionados con la resistencia antifúngica.
Además, se utilizaron términos de búsqueda y operadores booleanos tales como “(Opportunistic Mycoses) AND (Antifungal Resistance)” y “(Antifungals) AND (Resistance Genes)” para obtener artículos con información pertinente y relevante.
RESULTADOS
En la figura 1, se exhibe un diagrama de flujo que describe el procedimiento de selección según las normas PRISMA. La tabla 1 ofrece un análisis detallado de los resultados obtenidos en los estudios sobre resistencia antifúngica. El 58,33 % (n=7) de los estudios se centraron en la evaluación de la resistencia antifúngica, mientras que el 41,67 % (n=5) abordaron a profundidad otros aspectos relacionados, como mecanismos de acción y genética, junto con aspectos de diagnóstico basados en pruebas moleculares para la detección de resistencias.
El 66 % de los estudios se centraron en evaluar la resistencia en diversas especies de Candida spp., destacando C. auris. Este enfoque resalta la importancia de este género en la investigación sobre resistencia antifúngica. Además, tres estudios 16,7 % (n=2) abordaron la resistencia en Aspergillus spp., mientras que uno 8,3 % (n=1) se centró en Mucor circinelloides. Otro estudio 8,3 % (n=1) no solo investigó la resistencia de un patógeno, sino que se enfocó en diversos hongos, incluyendo Aspergillus spp., Penicillium, Rhizopus y Trichophyton mentagrophytes.
Entre los genes de resistencia encontrados, C. albicans es portador de los genes CDR1, CDR2, RTL3 y MAL2. C. parapsilosis presenta resistencia asociada al gen ERG11, mientras que, la resistencia en C. auris, estuvo relacionada con genes ERG3, ERG6 y ERG11. Además, en C. glabrata se identificaron los genes PDR1, FKS1 y FKS2, mientras que en C. lusitaniae se detectaron los genes MRR1, MFS7, FKS1, ERG3 y ERG4. En A. lentulus se encontró el gen CYP51A, y, por último, M. circinelloides presentó resistencia asociada con los genes PyrF y PyrG.
El fluconazol es el antifúngico más estudiado, seguido de la anfotericina B y el voriconazol. Otros medicamentos antifúngicos, entre ellos diferentes derivados de los azoles, olorofim, nistatina y 5-fluorocitosina, también fueron analizados en los estudios de interés. El 75 % (n=9) de los estudios mencionan aislamientos de origen no específico en sus análisis, mientras que el 25 % (n=3) de los estudios utilizaron aislamientos de muestras humanas de origen especificado.
Los métodos de laboratorio empleados en los estudios examinados para el análisis de resistencia e identificación de agentes fúngicos muestran variabilidad en su sensibilidad y especificidad tabla 2. SMRT (Single-Molecule Real-Time) sobresale como el método más sensible (90 %), mientras que la prueba de tubo germinal indica ser el método más específico (100 %) e indica una alta precisión en la identificación de C. albicans. Sin embargo, estos métodos pueden enfrentar interferencias, como contaminación de reactivos y condiciones de cultivo inadecuadas.
Por otro lado, técnicas como la reacción en cadena de la polimerasa (PCR) para identificación de genes de resistencia y la microdilución en caldo por medio RPMI, aunque sensibles, pueden verse afectadas por contaminaciones ambientales y cambios en la composición del medio, respectivamente. Mientras tanto, métodos como la espectrometría de masas de matriz asistida por láser (Vitek-2) y el cultivo en los agares mencionados en la tabla 2, aunque útiles, pueden experimentar problemas como la contaminación cruzada y presencia de elementos ajenos a la muestra.
Uno de los aspectos importantes que se debe tomar en cuenta al momento del análisis en el laboratorio clínico es la preanalítica tabla 3, los procedimientos de toma de muestra para el diagnóstico de infecciones fúngicas implican una serie de pasos para asegurar la efectividad en la detección, esto abarca, esterilización adecuada, obtención de tejido apropiado y uso de blanqueadores ópticos o tinta china según sea necesario; además, se aconseja la centrifugación de muestras antes de la microscopía para mejorar los resultados.
Las pruebas serológicas y moleculares son fundamentales para el diagnóstico en pacientes con riesgo de infección invasiva. Se resalta la importancia de analizar el suero en busca de antígeno criptocócico (CRAG) en pacientes con sospecha de meningitis criptocócica. Por otro lado, se recomienda realizar pruebas de galactomanano en suero y lavado broncoalveolar en pacientes con riesgo de aspergilosis invasiva.
Además, se indica que el estudio de β-D-glucano en suero se lleva a cabo en casos de riesgo de enfermedad fúngica invasiva en general, al igual que la PCR para Aspergillus. A su vez, se subraya la importancia de evaluar los anticuerpos contra Aspergillus, especialmente en pacientes con cavidades pulmonares y en situaciones de sospecha de aspergilosis broncopulmonar alérgica.
Para el estudio de infecciones fúngicas, es crucial determinar si el paciente presenta enfermedades subyacentes que puedan propiciar el desarrollo de micosis oportunistas. Se recomienda realizar un cultivo de líquidos, así como la identificación de levaduras y especies fúngicas específicas, además de pruebas de susceptibilidad para guiar el tratamiento adecuado.
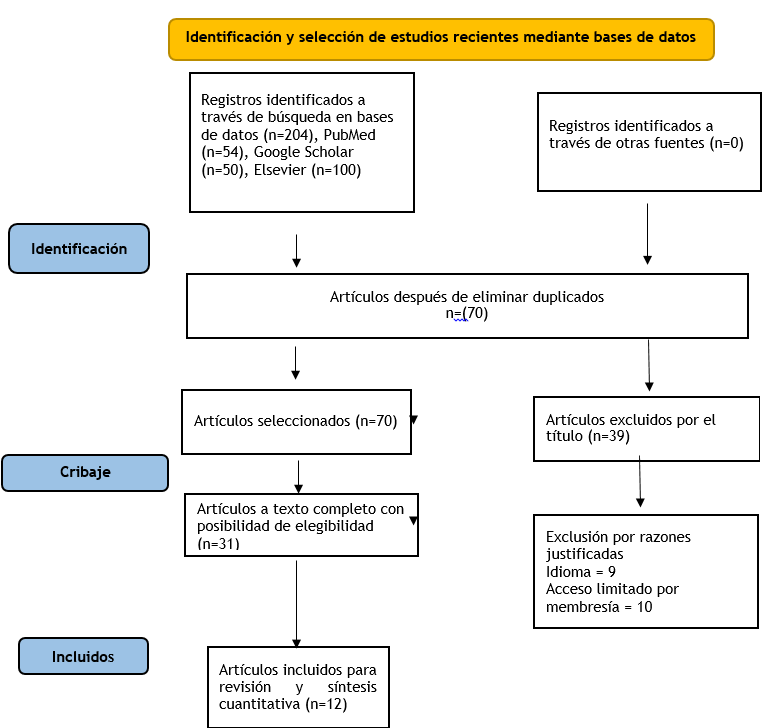
Figura 1. Flujograma PRISMA
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Tabla 2. Métodos utilizados en el laboratorio clínico para el cultivo de hongos y replicación de genes de resistencia |
|||
|
Pruebas de laboratorio |
Principio |
Muestra |
Interferencias |
|
Examen microscópico con KOH |
Incubación de muestra con diferentes concentraciones de KOH (20 % y 40 %), las células del tejido se digieren y los elementos fúngicos se vuelven visibles (S: 67 %; E: 87 %)(19) |
Muestras de piel y uñas Secreciones Líquidos corporales Tejido(19,20) |
Calidad de la muestra y la experiencia del observador pueden influir en la capacidad del KOH para detectar una infección de manera efectiva(20) |
|
Cultivo en Agar Papa Dextrosa |
Medio de cultivo no selectivo para el crecimiento de hongos y levaduras(21) |
Raspados de piel y uñas Secreciones(7,21) |
Aislamiento de especies fúngicas, el medio puede resultar en una alta tasa de contaminación, lo que puede interferir con el proceso de identificación(21) |
|
Cultivo en CHROMagar Cándida |
Detección de colonias de Cándida mediante un medio de cultivo selectivo que produce coloración específica: Verde manzana: C. albicans Azul opaco a púrpura con bordes rosa pálido: C. tropicalis Rosa pálido con superficies mate: C. krusei Blanco, brillante, de color rosa pálido a violeta: C. glabrata Rosa a morado: C. guilliermondii Rosa gris violeta: C. lusitaniae Blanco a rosa claro: C. famata Rosa pálido: C. parapsilosis(22) |
Secreciones Líquidos corporales(8,21) |
Identificación errónea de especies de Cándida por coloración similar(23) |
|
Cultivo en Agar Dextrosa Sabouraud |
Medio de cultivo selectivo, favorable para el aislamiento de hongos debido a su bajo pH, lo que dificulta el crecimiento bacteriano(24) |
Secreciones Líquidos corporales Raspados de córnea(8,24) |
Presencia de esporas de hongos ajenos a la muestra(24) |
|
Agar YPD |
Medio de cultivo enriquecido con extracto de levadura, peptona y dextrosa. Proporciona nutrientes esenciales para el crecimiento de levaduras(14,25) |
Sangre Raspados de piel(25) |
Contaminación cruzada del medio, errores en la preparación del agar(14) |
|
Prueba de tubo germinal |
Desarrollo de tubos germinales a partir de células fúngicas en condiciones de cultivo específicas. Método utilizado principalmente en la identificación de Cándida (S: 87 %; E: 100 %)(26) |
Secreciones Líquidos corporales(8) |
Falsos positivos por presencia de contaminantes; condiciones de cultivo inadecuadas(26) |
|
BMD-RPMI: microdilución en caldo utilizando medio RPMI |
Tubos suplementados con concentraciones de antifúngicos; permite evaluar la concentración mínima inhibitoria (MIC)(27) |
Secreciones Líquidos corporales(8) |
Cambios en la composición del medio durante la preparación, e incubación(27) |
|
Vitek-2 |
Espectrometría de masas de matriz asistida por láser para la identificación de microorganismos; pruebas de susceptibilidad a antifúngicos por métodos de difusión o dilución (S: 82 %; E: 97,2 %)(28) |
Secreciones Líquidos corporales Sangre(8,28)
|
Contaminación cruzada, errores en la preparación de la muestra, errores del equipo(28) |
|
PCR para genes de resistencia |
Técnica molecular que amplifica selectivamente regiones específicas de ADN. (S: 90 %; E: 72 %)(29) |
Secreciones Líquidos corporales Sangre(8,30) |
Contaminación ambiental durante el proceso, puede causar falsos positivos al igual que reactividad cruzada de los cebadores con ADN humano u otros hongos no objetivo(29) |
|
Secuenciación de SMRT (Single-Molecule Real-Time) |
Secuenciación de ADN que genera información de regiones variables y logra una identificación precisa de elementos fúngicos a nivel de especie (S: 90 %; E:94 %)(31,32) |
Tejido(32) |
Calidad de la muestra, contaminación de reactivos, interferencia de secuencias homólogas(31) |
|
Tabla 3. Recomendaciones de la toma de muestras para diagnóstico de infecciones fúngicas |
|
Toma de muestra para diagnóstico |
|
Microscopía y Tinciones Examinar muestras estériles y lavado broncoalveolar para detectar hongos mediante microscopía directa. Obtener tejido adecuado antes de la microscopía, especialmente en muestras no estériles. Usar blanqueadores ópticos en muestras de pacientes inmunocomprometidos. Realizar tinción con tinta china para líquido cefalorraquídeo si no hay prueba CRAG (Cryptococcal Antigen) disponible. Centrifugar muestras antes de la microscopía a 1000 go durante 10 minutos. Mezclar las muestras respiratorias para detección de Pneumocystis jirovecii.
Pruebas Serológicas y Moleculares Analizar suero para CRAG en pacientes con sospecha de meningitis criptocócica. Detectar galactomanano del suero en pacientes con alto riesgo de aspergilosis invasiva. Realizar prueba de galactomanano del lavado broncoalveolar (BAL) en pacientes con alto riesgo de aspergilosis invasiva. Detectar β-D-glucano en suero en pacientes con alto riesgo de enfermedad fúngica invasiva. Realizar PCR de suero para Aspergillus en pacientes con alto riesgo de enfermedad fúngica invasiva. Analizar suero para anticuerpos contra Aspergillus en pacientes con cavidades pulmonares de causa incierta. Realizar análisis de suero para IgE específica de Aspergillus en pacientes con sospecha de aspergilosis broncopulmonar alérgica.
Identificación Cultivar líquidos de broncoscopia en medios para hongos. Identificar levaduras en muestras de orina a nivel de especie para pacientes críticos o inmunocomprometidos. Identificar Aspergillus a nivel de complejo de especies para pacientes que recibirán tratamiento antifúngico. Identificar hongos de sitios estériles a nivel de complejo de especies y remitir a laboratorio especializado si es necesario. Realizar pruebas de susceptibilidad a antifúngicos para aislados de Candida spp y Aspergillus spp.
Solicitudes e Informes Clínicos Indicar si el paciente está inmunocomprometido en todas las solicitudes de pruebas. Retirar rápidamente dispositivos intravasculares después del diagnóstico de candidemia si es posible. Comunicar activamente resultados positivos a los médicos dentro de las 2 horas siguientes.
Monitoreo Terapéutico de Medicamentos No es necesario monitorizar anfotericina B o equinocandinas excepto en circunstancias excepcionales. Monitorizar itraconazol, voriconazol y posaconazol, especialmente voriconazol.(33) |
DISCUSIÓN
La resistencia antifúngica resulta de mutaciones en genes clave que brindan a los hongos protección contra fármacos específicos, este problema, aunque no se ha considerado completamente, es significativo en pacientes con otras enfermedades que pueden verse afectadas por micosis oportunistas. La resistencia de los hongos a los medicamentos representa un desafío significativo debido a la escasa variedad de tratamientos disponibles, lo que conlleva al agotamiento rápido de opciones terapéuticas cuando los pacientes desarrollan resistencia.(34)
Según varios estudios, el género Candida spp. ha sido objeto de amplia investigación debido a su creciente resistencia, como lo demuestra C. auris, que exhibe una significativa resistencia a la anfotericina B, alcanzando tasas hasta del 60 % en aislamientos clínicos.(17) Además, varias investigaciones han identificado diversos genes asociados con resistencia en C. albicans tabla 1.
El fluconazol es el antifúngico más utilizado para estas infecciones, ya que inhibe la enzima lanosterol desmetilasa del citocromo P450, codificada por el gen ERG11, implicado en la vía de biosíntesis del ergosterol; muchos aislamientos clínicos de C. albicans muestran una sobreexpresión de ERG11, el blanco de los azoles.(35)
Además del ERG11, se han identificado elementos de resistencia a polienos y azoles, asociados con mutaciones en genes como ERG3 y ERG4, que codifican enzimas implicadas en la síntesis de ergosterol. Por otro lado, ERG6, responsable de la conversión de zimosterol o lanosterol en precursores alternativos de ergosterol, también está implicado en la resistencia en especies de Mucor spp.(36,37) Además, los genes PyrF y PyrG relacionados con la sensibilidad de Mucor circinelloides al 5-FOA, mediante la conversión del ácido 5-fluoroorótico (5-FOA) en su forma tóxica activa, cuando sufren epimutaciones pueden generar el silenciamiento de estos genes, confiriendo resistencia a este antifúngico.(14)
El estudio, enfocado en Aspergillus lentulus., aislado del esputo de un paciente con enfisema obstructivo crónico(13), detectó mutaciones en CYP51A, que codifica para la enzima en la esterol-14α-desmetilasa, generando alteraciones en la síntesis de ergosterol en la membrana celular fúngica.(38) Los patógenos fúngicos de este género viven en ambientes interiores y exteriores, por lo que, Sewell y colaboradores(39) analizaron muestras directas del suelo para identificar resistencias de diversas cepas de Apergillus spp., encontrando la presencia de los genes CYP51A y CSP1, relacionados con resistencia a ciertos azoles.
Además, se han identificado genes relacionados con transportadores ABC de resistencia multidroga tabla 1, agrupados en dos subfamilias principales, PDR (AtrB, AtrF CDR1 y CDR2), y MDR (MDR1, MDR2 Y MDR3), así como genes vinculados al transportador de multidrogas MFS; estos genes codifican proteínas transportadoras que desempeñan un papel en la expulsión activa de los fármacos antifúngicos fuera de la célula del hongo, lo que disminuye la eficacia terapéutica.(13)
Las cepas QDRKO exhiben una expresión diferencial de genes relacionados con el transporte de glucosa, lo que resulta en una acumulación intracelular aumentada de glucosa y glicerol, además, se observa una mayor resistencia a los fármacos azoles y una formación mejorada de formas de hifas, lo que sugiere un papel importante de los genes QDR en la virulencia y patogenicidad de C. albicans.(18)
En el ámbito del diagnóstico de infecciones fúngicas, existe una amplia gama de pruebas disponibles, que van desde las más simples y comúnmente empleadas, como el examen microscópico con KOH, hasta técnicas moleculares altamente sofisticadas. A pesar de esta variedad, todas las pruebas de laboratorio pueden conllevar riesgos de errores en la identificación de hongos patógenos y sus resistencias, sin embargo, métodos como la secuenciación en tiempo real, PCR, entre otros, ofrecen una sensibilidad y especificidad superiores, lo que garantiza una mayor precisión en los resultados diagnósticos.
Es esencial considerar la relación entre el costo y la complejidad de las pruebas utilizadas. En este sentido, se destaca el uso de medios de cultivo para agentes fúngicos debido a su practicidad. Sin embargo, estos medios pueden ser propensos a interferencias; por ejemplo, los agares Papa Dextrosa, Dextrosa Sabouraud y YPD pueden experimentar contaminación y variabilidad en su composición, lo que afecta el proceso de identificación de especies fúngicas.(24)
Otro medio de cultivo de uso frecuente, pero con un costo superior, es el CHROMagar específico para Candida, utilizado desde los años 90(40), el cual permite una rápida identificación de diversas especies de este género mediante una coloración diferencial específica. En contraste, otros agares no permiten distinguir la especie de Candida en estudio, lo que hace que este medio sea altamente específico; no obstante, los errores analíticos pueden derivar de una interpretación incorrecta de la coloración.(23)
A pesar de contar con una especificidad del 100 %, la prueba de Tubo Germinal muestra una sensibilidad del 87,1 %, lo que sugiere una capacidad moderada para detectar la presencia de células fúngicas en muestras en general.(26) Sin embargo, su atractivo radica en su costo accesible en comparación con los métodos fenotípicos y PCR, cuyos costos por aislamiento resultan considerablemente elevados; en contraste, la prueba de Tubo Germinal se destaca por su costo relativamente insignificante(41), lo que la convierte en una opción frecuentemente utilizada.
Por otro lado, la PCR para genes de resistencia y la secuenciación de SMRT pueden enfrentar desafíos relacionados con la contaminación ambiental y la interferencia de secuencias homólogas, lo que puede afectar la precisión de los resultados.(29,31) En comparación, el medio RPMI también permite la evaluación de la resistencia, mediante el análisis de la concentración mínima inhibitoria con un riesgo de error por la preparación del medio como puede darse en métodos de agares.(27)
En términos de precisión en métodos de biología molecular, la secuenciación SMRT demostró ser el más sensible y específico (S: 90 %; E: 94 %), seguido de Vitek-2 (S: 82 %; E: 97,2 %) y PCR (S: 90 %; E: 72 %).(28,29,32) A pesar de la alta sensibilidad y especificidad de estos métodos, el examen microscópico con KOH, debido a su simplicidad y bajo, sigue siendo el método preferido para la diferenciación de elementos fúngicos(19) y la microdilución en caldo BMD-RMPI es el método preferido para la evaluación de resistencia debido a su accesibilidad.(27)
Al mismo tiempo se deben considerar aspectos de la fase preanalítica, y los exámenes que deben considerarse según el caso y el tipo de sospecha de infección fúngica tabla 3; al sospechar de infección fúngica invasiva es importante utilizar pruebas de diagnóstico no basadas en cultivos, como galactomanano, beta-d-glucano, tecnología de flujo lateral, resonancia magnética T2 y PCR, en las cuales, los resultados positivos incluyen el diagnóstico cuando la prevalencia de la enfermedad es alta.(42)
CONCLUSIONES
La identificación precoz de la resistencia antifúngica emerge como un pilar fundamental en la mitigación de su impacto en la salud pública, lo cual implica una inversión significativa en recursos, tanto en programas de vigilancia epidemiológica como en políticas preventivas; es esencial promover el monitoreo regular de la susceptibilidad a los antifúngicos y fomentar prácticas responsables de su uso en diversos entornos clínicos.
Se ha observado una preocupante tendencia hacia la resistencia antifúngica en especies clínicamente relevantes, como Candida spp. y Aspergillus spp., fenómeno que resalta la urgencia de desarrollar nuevas estrategias terapéuticas dirigidas específicamente a los mecanismos de resistencia identificados, incluyendo la inhibición de genes asociados a la resistencia y la exploración de terapias combinadas que reduzcan la emergencia de cepas resistentes.
La disparidad en los métodos de laboratorio para detectar resistencia antifúngica enfatiza la necesidad de estandarizar protocolos de diagnóstico; la adopción de técnicas altamente sensibles y específicas como método diagnóstico primario podría agilizar la selección de tratamientos efectivos y disminuir la propagación de la resistencia
REFERENCIAS BIBLIOGRÁFICAS
1. Wiederhold NP. Emerging Fungal Infections: New Species, New Names, and Antifungal Resistance. Clin Chem. 2021;68(1):83–90.
2. Kuchi Bhotla H, Balasubramanian B, Meyyazhagan A, Pushparaj K, Easwaran M, Pappusamy M, et al. Opportunistic mycoses in COVID-19 patients/survivors: Epidemic inside a pandemic. J Infect Public Health. 2021;14(11):1720–6.
3. Kean R, Ramage G. Combined Antifungal Resistance and Biofilm Tolerance: the Global Threat of Candida auris. mSphere. 2019;4(4).
4. Rogers TR, Verweij PE, Castanheira M, Dannaoui E, White PL, Arendrup MC, et al. Molecular mechanisms of acquired antifungal drug resistance in principal fungal pathogens and EUCAST guidance for their laboratory detection and clinical implications. Journal of Antimicrobial Chemotherapy. 2022;77(8):2053–73.
5. Ivanov M, Ćirić A, Stojković D. Emerging Antifungal Targets and Strategies. Int J Mol Sci. 2022;23(5):2756.
6. Qin Y, Wang J, Lv Q, Han B. Recent Progress in Research on Mitochondrion-Targeted Antifungal Drugs: a Review. Antimicrob Agents Chemother. 2023;67(6).
7. Bazira J, Nalumaga P, Tuhamize B, Orishaba F, Frederickson W, Taseera K. Antifungal Susceptibility of Pathogenic Moulds isolated from patients at Tertiary Hospital in South western Uganda. 2023.
8. Miruka H, Omwenga E, Stanslaus M, Silas A. Incidence, antifungal resistance properties, and virulence traits of Candida species isolated from HIV/AIDS Patients from the hospital system in Kenya. 2022.
9. Zaragoza O, Alcázar L, Trevijano N. Global emergence of resistance to fluconazole and voriconazole in Candida parapsilosis in tertiary hospitals in Spain during the COVID-19 pandemic. MedRxiv. 2022.
10. Rasouli S, Shankarnarayan S, Galon C, Charlebois DA. Identification and Elimination of Antifungal Tolerance in Candida auris. bioRxiv. 2022.
11. Valero C, Mcmanus M, Ashvatti A, Tindale J, Gago S, Bromley M. Reversing olorofim activity reveals multifactorial drug tolerance in Aspergillus fumigatus. bioRxiv. 2023.
12. Arastehfar A, Daneshnia F, Hovhannisyan H, Cabrera N, Ilkit M, Desai J, et al. A multidimensional assessment of in-host fitness costs of drug resistance in the opportunistic fungal pathogen Candida glabrata. bioRxiv. 2023.
13. Wang X, Yusufu A, Hasimu H, Abliz P. Whole genome sequencing of newly emerged fungal pathogen Aspergillus lentulus and its Azole resistance gene prediction. bioRxiv. 2023.
14. Chang Z, Billmyre RB, Chan Lee S, Heitman J. Broad antifungal resistance mediated by RNAi-dependent epimutation in the basal human fungal pathogen Mucor circinelloides. bioRxiv. 2019.
15. Kannan A, Asner SA, Trachsel E, Kelly S, Parker J, Sanglard D. Comparative genomics for the elucidation of multidrug resistance (MDR) in Candida lusitaniae. bioRxiv. 2019.
16. Luan Q, Zou P, Liu J, Li P. RETRACTED: Antifungal activity, synergism with fluconazole or amphotericin B and potential mechanism of direct current against Candida albicans biofilms and persisters. Res Sq. 2023.
17. Carolus H, Sofras D, Boccarella G, Sephton-Clark P, Lobo Romero C, Vergauwen R, et al. Acquired amphotericin B resistance and fitness trade-off compensation in Candida auris. Res Sq. 2023.
18. Qadri H, Haseeb Shah A, Ahmad Mir M, Fazal Qureshi M, Prasad R. Quinidine drug resistance transporter knockout Candida cells modulate glucose transporter expression and accumulate metabolites leading to enhanced azole drug resistance. Fungal Genetics and Biology. 2022.
19. Bhabhor U, Mistry Y, Mullan S. Evaluation of Sensitivity and Specificity of Direct Microscopical Examination of Suspected Mucor Mycosis Samples by Potassium Hydroxide (KOH) during Covid-19 Pandemic Era. Adv Infect Dis. 2022;12(04):776–80.
20. Dass SM, Vinayaraj E V., Pavavni K, Pallam A, Rao MS. Comparison of KOH, Calcofluor White and Fungal Culture for Diagnosing Fungal Onychomycosis in an Urban Teaching Hospital, Hyderabad. Indian Journal of Microbiology Research. 2015;2(3):148.
21. Park HJ, Kim JH, Ryu JW. Differences in the accuracy of potato dextrose agar and CHROMagar media in diagnosing oral candidiasis. Oral Biology Research. 2021;45(3):119–24.
22. Nadeem SG, Hakim ST, Kazmi SU. Use of CHROMagar Candida for the presumptive identification of Candida species directly from clinical specimens in resource-limited settings. Libyan Journal of Medicine. 2010;5(1):2144.
23. de Jong AW, Dieleman C, Carbia M, Mohd Tap R, Hagen F. Performance of Two Novel Chromogenic Media for the Identification of Multidrug-Resistant Candida auris Compared with Other Commercially Available Formulations. J Clin Microbiol. 2021;59(4).
24. Das S, Sharma S, Kar S, Sahu S, Samal B, Mallick A. Is inclusion of Sabouraud dextrose agar essential for the laboratory diagnosis of fungal keratitis? Indian J Ophthalmol. 2010;58(4):281.
25. Das S, Singh S, Tawde Y, Chakrabarti A, Rudramurthy SM, Kaur H, et al. A Selective Medium for Isolation and Detection of Candida auris, an Emerging Pathogen. J Clin Microbiol. 2021;59(2).
26. Sheppard DC, Locas MC, Restieri C, Laverdiere M. Utility of the Germ Tube Test for Direct Identification of Candida albicans from Positive Blood Culture Bottles. J Clin Microbiol. 2008;46(10):3508–9.
27. Shields RK, Nguyen MH, Press EG, Updike CL, Clancy CJ. Caspofungin MICs Correlate with Treatment Outcomes among Patients with Candida glabrata Invasive Candidiasis and Prior Echinocandin Exposure. Antimicrob Agents Chemother. 2013;57(8):3528–35.
28. Ruiz de Alegría Puig C, Agüero-Balbín J, Fernández-Mazarrasa C, Martínez-Martínez L. Evaluation of the Vitek-MSTM system in the identification of Candida isolates from bloodstream infections. Rev Iberoam Micol. 2018;35(3):130–3.
29. Garcia-Effron G. Molecular Markers of Antifungal Resistance: Potential Uses in Routine Practice and Future Perspectives. Journal of Fungi. 2021;7(3):197.
30. Khot PD, Fredricks DN. PCR-based diagnosis of human fungal infections. Expert Rev Anti Infect Ther. 2009;7(10):1201–21.
31. Long N, Liu J, Liu J, Li Y, Hou Y, Liao X, et al. Single-Molecule Real-Time Sequencing to Explore the Mycobiome Diversity in Malt. Microbiol Spectr. 2022;10(5).
32. Tse OYO, Jiang P, Cheng SH, Peng W, Shang H, Wong J, et al. Genome-wide detection of cytosine methylation by single molecule real-time sequencing. Proceedings of the National Academy of Sciences. 2021;118(5).
33. Schelenz S, Barnes RA, Barton RC, Cleverley JR, Lucas SB, Kibbler CC, et al. British Society for Medical Mycology best practice recommendations for the diagnosis of serious fungal diseases. Lancet Infect Dis. 2015;15(4):461–74.
34. Vitiello A, Ferrara F, Boccellino M, Ponzo A, Cimmino C, Comberiati E, et al. Antifungal Drug Resistance: An Emergent Health Threat. Biomedicines. 2023;11(4):1063.
35. Berkow E, Lockhart S. Fluconazole resistance in Candida species: a current perspective. Infect Drug Resist. 2017; Volume 10:237–45.
36. Lotfali E, Ghajari A, Kordbacheh P, Zaini F, Mirhendi H, Mohammadi R, et al. Regulation of ERG3, ERG6, and ERG11 Genes in Antifungal-Resistant isolates of Candida parapsilosis. Iran Biomed J. 2017;21(4):275–81.
37. Navarro MI, Pérez C, Parker J, Kelly S, Heitman J. Alternative ergosterol biosynthetic pathways confer antifungal drug resistance in the human pathogens within the Mucor species complex. bioRxiv. 2023.
38. Bottery MJ, Rhijn N van, Chown H, Rhodes JL, Celia BN, Brewer MT, et al. Elevated mutation rates in the multi-azole resistant Aspergillus fumigatus clade drives rapid evolution of antifungal resistance. bioRxiv. 2023.
39. Sewell TR, Zhang Y, Brackin AP, Shelton JMG, Rhodes J, Fisher MC. Elevated Prevalence of Azole-Resistant Aspergillus fumigatus in Urban versus Rural Environments in the United Kingdom. Antimicrob Agents Chemother. 2019;63(9).
40. Bouchara JP, Declerck P, Cimon B, Planchenault C, de Gentile L, Chabasse D. Routine use of CHROMagar Candida medium for presumptive identification of Candida yeast species and detection of mixed fungal populations. Clinical Microbiology and Infection. 1996;2(3):202–8.
41. Jan A, Bashir G, Qadir R, Fomda BA, Sofia, Yaqoob-Hakak A. Modified Germ Tube Test: A Rapid Test for Differentiation of Candida albicans from Candida dubliniensis. International Journal of Contemporary Medical Research. 2018;5(3).
42. Patterson TF, Donnelly JP. New Concepts in Diagnostics for Invasive Mycoses: Non-Culture-Based Methodologies. Journal of Fungi. 2019;5(1):9.
FINANCIACIÓN
Ninguna
CONFLICTO DE INTERESES
Los autores declaran que no existe conflicto de intereses.
CONTRIBUCIÓN DE AUTORÍA
Conceptualización: Natalia Raquel Aguiar Muñoz, Carlos Fernando Yauli Flores.
Investigación: Natalia Raquel Aguiar Muñoz.
Metodología: Natalia Raquel Aguiar Muñoz, Carlos Fernando Yauli Flores.
Administración del proyecto: Natalia Raquel Aguiar Muñoz.
Supervisión: Carlos Fernando Yauli Flores.
Redacción – borrador original: Natalia Raquel Aguiar Muñoz, Carlos Fernando Yauli Flores.
Redacción – revisión y edición: Natalia Raquel Aguiar Muñoz, Carlos Fernando Yauli Flores.